Valeurs manquantes et factorisation de matrices#
Links: notebook, html, PDF, python, slides, GitHub
Réflexion autour des valeur manquantes et de la factorisation de matrice positive.
from jyquickhelper import add_notebook_menu
add_notebook_menu()
%matplotlib inline
Matrice à coefficients aléatoires#
On étudie la factorisation d’une matrice à coefficients tout à fait
aléatoires qui suivent une loi uniforme sur l’intervalle .
Essayons sur une petite matrice :
from numpy.random import rand
M = rand(3, 3)
M
array([[ 0.05119593, 0.43722929, 0.9290821 ],
[ 0.4588466 , 0.14187813, 0.23762633],
[ 0.9768084 , 0.47674026, 0.79044526]])
from sklearn.decomposition import NMF
mf = NMF(1)
mf.fit_transform(M)
array([[ 0.67825803],
[ 0.38030919],
[ 1.02295362]])
La matrice précédente est la matrice dans le produit
, la matrice qui suit est
.
mf.components_
array([[ 0.73190904, 0.50765757, 0.92611883]])
mf.reconstruction_err_ / (M.shape[0] * M.shape[1])
0.07236890712696428
On recalcule l’erreur :
d = M - mf.fit_transform(M) @ mf.components_
a = d.ravel()
e = a @ a.T
e ** 0.5 / (M.shape[0] * M.shape[1])
0.072368907126964283
e.ravel()
array([ 0.42421796])
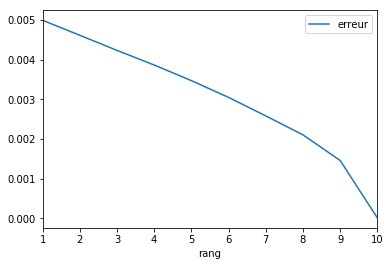
Et maintenant sur une grande et plus nécessairement carrée :
M = rand(300, 10)
mf = NMF(1)
mf.fit_transform(M)
mf.reconstruction_err_ / (M.shape[0] * M.shape[1])
0.004996164872801101
L’erreur est la même :
errs = []
rangs = list(range(1, 11))
for k in rangs:
mf = NMF(k)
mf.fit_transform(M)
e = mf.reconstruction_err_ / (M.shape[0] * M.shape[1])
errs.append(e)
import pandas
df = pandas.DataFrame(dict(rang=rangs, erreur=errs))
df.plot(x="rang", y="erreur")
<matplotlib.axes._subplots.AxesSubplot at 0x199924d8668>
Matrice avec des vecteurs colonnes corrélés#
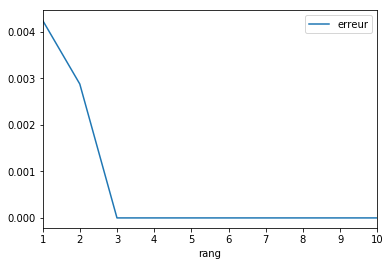
Supposons maintenant que la matrice précédente est de rang 3.
Pour s’en assurer, on tire une matrice aléalatoire avec 3 vecteurs
colonnes et on réplique des colonnes jusqu’à la dimension souhaitée.
from numpy import hstack
M = rand(300, 3)
M = hstack([M, M, M, M[:,:1]])
M.shape
(300, 10)
errs = []
rangs = list(range(1, 11))
for k in rangs:
mf = NMF(k)
mf.fit_transform(M)
e = mf.reconstruction_err_ / (M.shape[0] * M.shape[1])
errs.append(e)
import pandas
df = pandas.DataFrame(dict(rang=rangs, erreur=errs))
df.plot(x="rang", y="erreur")
<matplotlib.axes._subplots.AxesSubplot at 0x199925d6630>
On essaye à nouveausur une matrice un peu plus petite.
M = rand(3, 2)
M = hstack([M, M[:,:1]])
M
array([[ 0.27190312, 0.6497563 , 0.27190312],
[ 0.44853292, 0.87097224, 0.44853292],
[ 0.29424835, 0.65106952, 0.29424835]])
mf = NMF(2)
mf.fit_transform(M)
array([[ 0.61835197, 0. ],
[ 0.82887888, 0.29866219],
[ 0.61960446, 0.07743224]])
mf.components_
array([[ 0.43972536, 1.05078419, 0.43972536],
[ 0.28143493, 0. , 0.28143493]])
La dernière colonne est identique à la première.
Matrice identité#
Et maintenant si la matrice est la matrice identité, que se
passe-t-il ?
from numpy import identity
M = identity(3)
M
array([[ 1., 0., 0.],
[ 0., 1., 0.],
[ 0., 0., 1.]])
mf = NMF(1)
mf.fit_transform(M)
array([[ 0.],
[ 1.],
[ 0.]])
mf.components_
array([[ 0., 1., 0.]])
mf.reconstruction_err_ ** 2
2.0000000000000004
On essaye avec .
mf = NMF(2)
mf.fit_transform(M)
array([[ 0. , 0. ],
[ 0. , 1.03940448],
[ 0.95521772, 0. ]])
mf.components_
array([[ 0. , 0. , 1.04688175],
[ 0. , 0.96208937, 0. ]])
mf.reconstruction_err_ ** 2
1.0
Avec des vecteurs normés et indépendants (formant donc une base de
l’espace vectoriel), l’algorithme aboutit à une matrice égale
au
premiers vecteurs et oublie les autres.
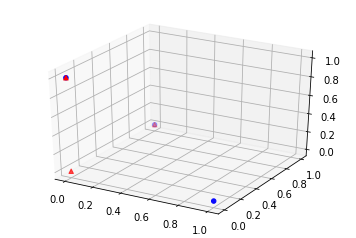
Matrice identité et représentation spatiale#
Pour comprendre un peu mieux ce dernier exemple, il est utile de chercher d’autres solutions dont l’erreur est équivalente.
def erreur_mf(M, W, H):
d = M - W @ H
a = d.ravel()
e = a @ a.T
e ** 0.5 / (M.shape[0] * M.shape[1])
return e
M = identity(3)
mf = NMF(2)
W = mf.fit_transform(M)
H = mf.components_
erreur_mf(M, W, H)
1.0
W
array([[ 0. , 0. ],
[ 0.9703523 , 0. ],
[ 0. , 1.02721047]])
H
array([[ 0. , 1.03055354, 0. ],
[ 0. , 0. , 0.97351032]])
W @ H
array([[ 0., 0., 0.],
[ 0., 1., 0.],
[ 0., 0., 1.]])
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
wh = W @ H
ax.scatter(M[:,0], M[:,1], M[:,2], c='b', marker='o', s=20)
ax.scatter(wh[:,0], wh[:,1], wh[:,2], c='r', marker='^')
<mpl_toolkits.mplot3d.art3d.Path3DCollection at 0x19992d2a5c0>
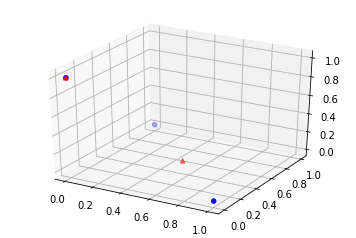
Et si on pose maintenant :
import numpy
W = numpy.array([[0.5, 0.5, 0], [0, 0, 1]]).T
H = numpy.array([[1, 1, 0], [0.0, 0.0, 1.0]])
W
array([[ 0.5, 0. ],
[ 0.5, 0. ],
[ 0. , 1. ]])
H
array([[ 1., 1., 0.],
[ 0., 0., 1.]])
W @ H
array([[ 0.5, 0.5, 0. ],
[ 0.5, 0.5, 0. ],
[ 0. , 0. , 1. ]])
erreur_mf(M, W, H)
1.0
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
wh = W @ H
ax.scatter(M[:,0], M[:,1], M[:,2], c='b', marker='o', s=20)
ax.scatter(wh[:,0], wh[:,1], wh[:,2], c='r', marker='^')
<mpl_toolkits.mplot3d.art3d.Path3DCollection at 0x19992a2e5f8>
On peut voir la matrice comme un ensemble de
points
dans un espace vectoriel. La matrice
est un ensemble de
points dans le même espace. La matrice
, de rang
est une approximation de cet ensemble dans le même espace,
c’est aussi
combinaisons linéaires de
points de
façon à former
points les plus proches proches de
points de la matrice
.