Saisonnalités, changement de régime, COVID en France#
Links: notebook, html, PDF, python, slides, GitHub
On s’intéresse aux séries temporelles de l’épidémie du COVID en France récupérées depuis data.gouv.fr : Chiffres-clés concernant l’épidémie de COVID19 en France.
from jyquickhelper import add_notebook_menu
add_notebook_menu()
%matplotlib inline
Données#
from pandas import DataFrame, read_csv, to_datetime
df = read_csv("https://www.data.gouv.fr/en/datasets/r/0b66ca39-1623-4d9c-83ad-5434b7f9e2a4")
df.tail()
c:python387_x64libsite-packagesIPythoncoreinteractiveshell.py:3146: DtypeWarning: Columns (17,18) have mixed types.Specify dtype option on import or set low_memory=False. has_raised = await self.run_ast_nodes(code_ast.body, cell_name,
| date | granularite | maille_code | maille_nom | cas_confirmes | cas_ehpad | cas_confirmes_ehpad | cas_possibles_ehpad | deces | deces_ehpad | reanimation | hospitalises | nouvelles_hospitalisations | nouvelles_reanimations | gueris | depistes | source_nom | source_url | source_archive | source_type | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 43080 | 2021-02-10 | region | REG-75 | Nouvelle-Aquitaine | NaN | NaN | NaN | NaN | 2510.0 | NaN | 213.0 | 1524.0 | 78.0 | 20.0 | 10227.0 | NaN | OpenCOVID19-fr | NaN | NaN | opencovid19-fr |
| 43081 | 2021-02-10 | region | REG-76 | Occitanie | NaN | NaN | NaN | NaN | 2882.0 | NaN | 276.0 | 1886.0 | 121.0 | 25.0 | 12801.0 | NaN | OpenCOVID19-fr | NaN | NaN | opencovid19-fr |
| 43082 | 2021-02-10 | region | REG-84 | Auvergne-Rhône-Alpes | NaN | NaN | NaN | NaN | 8453.0 | NaN | 400.0 | 3699.0 | 213.0 | 42.0 | 34579.0 | NaN | OpenCOVID19-fr | NaN | NaN | opencovid19-fr |
| 43083 | 2021-02-10 | region | REG-93 | Provence-Alpes-Côte d'Azur | NaN | NaN | NaN | NaN | 5010.0 | NaN | 438.0 | 3581.0 | 240.0 | 33.0 | 24080.0 | NaN | OpenCOVID19-fr | NaN | NaN | opencovid19-fr |
| 43084 | 2021-02-10 | region | REG-94 | Corse | NaN | NaN | NaN | NaN | 133.0 | NaN | 7.0 | 58.0 | 4.0 | 3.0 | 587.0 | NaN | OpenCOVID19-fr | NaN | NaN | opencovid19-fr |
# Il y a une date 2020-11_11
df['date'] = to_datetime(df['date'].apply(lambda s: s.replace("_", "-")))
gr = df[["date", "cas_confirmes"]].groupby("date").sum().sort_index()
gr.tail()
| cas_confirmes | |
|---|---|
| date | |
| 2021-02-06 | 3317333.0 |
| 2021-02-07 | 3337048.0 |
| 2021-02-08 | 3341365.0 |
| 2021-02-09 | 3360235.0 |
| 2021-02-10 | 3385622.0 |
gr['cas_confirmes'].plot(figsize=(14, 4), grid=True, title="Cas positifs");
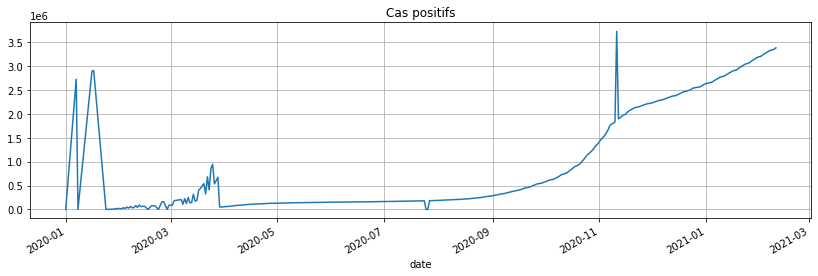
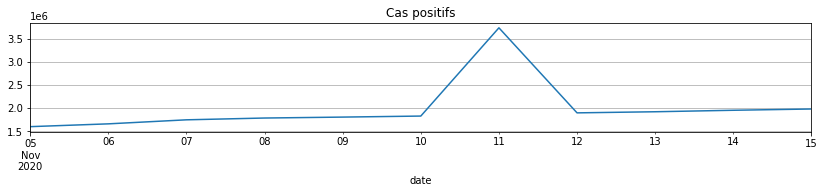
Il y a des petits problèmes de données… On corrige de façon automatique ou à la main… Etant donné que les données n’étaient pas fiables avant juin 2020 car pas assez de tests, on commence la série en septembre 2020. Il ne reste donc plus qu’une date un peu problématique, le 11 novembre.
from datetime import datetime
gr.loc[(gr.index >= datetime(2020, 11, 5)) & (gr.index <= datetime(2020, 11, 15)), 'cas_confirmes'].plot(
figsize=(14, 2), grid=True, title="Cas positifs");
gr.loc[datetime(2020, 11, 11), 'cas_confirmes'] = (
gr.loc[datetime(2020, 11, 10), 'cas_confirmes'] + gr.loc[datetime(2020, 11, 12), 'cas_confirmes']) / 2
gr.loc[gr.index >= datetime(2020, 9, 1), 'cas_confirmes'].plot(figsize=(14, 4), grid=True, title="Cas positifs");
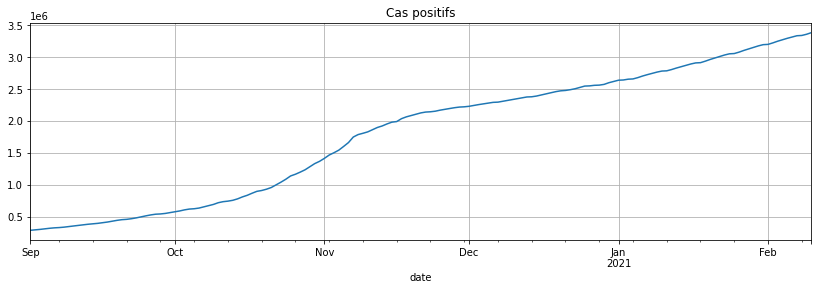
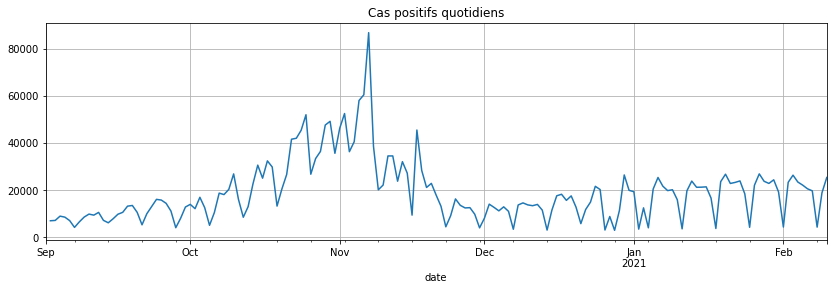
C’est mieux. On regarde la série différenciée :
covsept = gr.loc[gr.index >= datetime(2020, 9, 1), 'cas_confirmes']
covsept.diff().plot(figsize=(14, 4), grid=True, title="Cas positifs quotidiens");
Changements de régimes#
fbprophet quand ça marche#
from fbprophet import Prophet
from fbprophet.plot import add_changepoints_to_plot
try:
m = Prophet(changepoint_prior_scale=0.5)
fig = m.plot(covsept)
a = add_changepoints_to_plot(fig.gca(), m, covsept)
except AttributeError as e:
print(e)
# Sur Windows, fbprophet est compliqué à installer.
# J'ai abandonné. C'est plus simple sous Linux.
Importing plotly failed. Interactive plots will not work.
'Prophet' object has no attribute 'stan_backend'
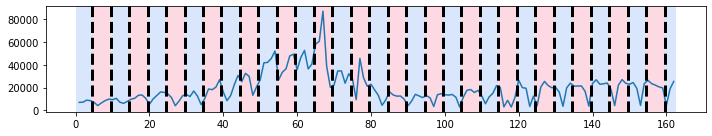
On passe à un autre module. Il compare localement la moyenne de la série à la série elle-même. Sur ce type de série avec une périodicité, ça ne marche pas très bien.
Ecart à la moyenne#
import ruptures
signal = covsept.diff().values
algo = ruptures.Pelt(model="rbf").fit(signal)
result = algo.predict(pen=10)
ruptures.display(signal, result, result);
import numpy
signal = covsept.diff().values
X = numpy.arange(signal.shape[0]).reshape(-1, 1).astype(signal.dtype)[1:]
y = signal[1:]
X.shape, y.shape
((162, 1), (162,))
Cuisine maison#
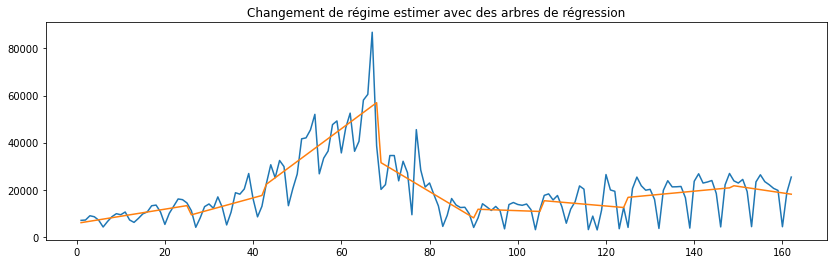
Donc je suis finalement passé à quelque chose que j’ai codé : un arbre de régression mais donc chaque feuille est une régression linéaire et non une constante. Ca manque un peu de théorie mais rien n’empêche d’être un peu inventif :PiecewiseTreeRegressor.
from mlinsights.mlmodel import PiecewiseTreeRegressor
ptr = PiecewiseTreeRegressor(min_samples_leaf=14)
ptr.fit(X, y)
C:xavierdupre__home_github_forkscikit-learnsklearntree_classes.py:335: FutureWarning: The parameter 'X_idx_sorted' is deprecated and has no effect. It will be removed in 1.1 (renaming of 0.26). You can suppress this warning by not passing any value to the 'X_idx_sorted' parameter. warnings.warn(
PiecewiseTreeRegressor(min_samples_leaf=14)
import matplotlib.pyplot as plt
fig, ax = plt.subplots(1, 1, figsize=(14, 4))
ax.plot(X, y)
ax.plot(X, ptr.predict(X))
ax.set_title("Changement de régime estimer avec des arbres de régression");
L’épidémie progresse de façon explonentielle. Il vaudrait sans doute mieux étudier le logarithme de la série différenciée.
ptrl = PiecewiseTreeRegressor(min_samples_leaf=14)
ly = numpy.log(y)
ptrl.fit(X, ly)
fig, ax = plt.subplots(1, 1, figsize=(14, 4))
ax.plot(X, ly)
ax.plot(X, ptrl.predict(X))
ax.set_title("Changement de régime estimer avec des arbres de régression"
"\nversion logarithmique");
C:xavierdupre__home_github_forkscikit-learnsklearntree_classes.py:335: FutureWarning: The parameter 'X_idx_sorted' is deprecated and has no effect. It will be removed in 1.1 (renaming of 0.26). You can suppress this warning by not passing any value to the 'X_idx_sorted' parameter. warnings.warn(
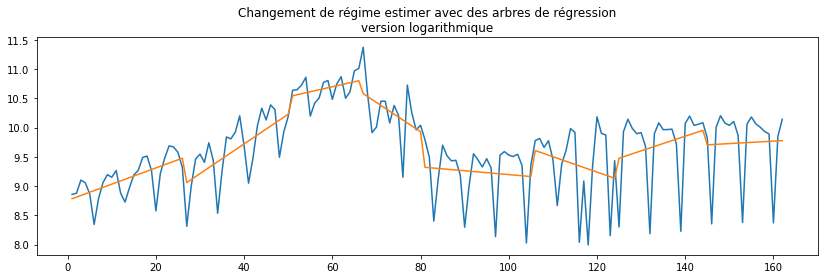
C’est un petit peu mieux. Cette méthode s’applique surtout à la tendance des séries contrairement à la méthode suivante plus approprié pour segmenter les résidus.
Les modules classiques#
Saisonnalité#
Saisonnalité et moyennes mobiles#
from statsmodels.tsa.seasonal import seasonal_decompose
res = seasonal_decompose(ly, freq=7)
res.plot();
<ipython-input-21-96d157ac9bae>:2: FutureWarning: the 'freq'' keyword is deprecated, use 'period' instead
res = seasonal_decompose(ly, freq=7)
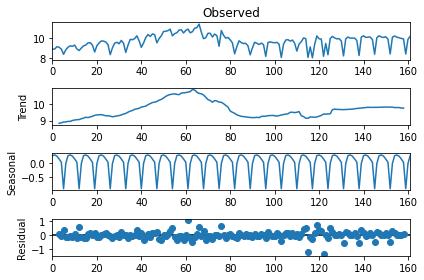
res.resid[:10]
array([ nan, nan, nan, 0.07542526, -0.02752109,
0.3475441 , -0.11152892, -0.15329366, -0.04130195, -0.09907971])
Quelque chose qui n’a pas beaucoup de sens#
model = GaussianHMM(n_components=3, covariance_type="diag", n_iter=1000)
ry = res.trend[3:-3] # on enlève les nan
model.fit(ry.reshape(-1, 1))
hidden_states = model.predict(ry.reshape(-1, 1))
set(hidden_states)
{0, 1, 2}
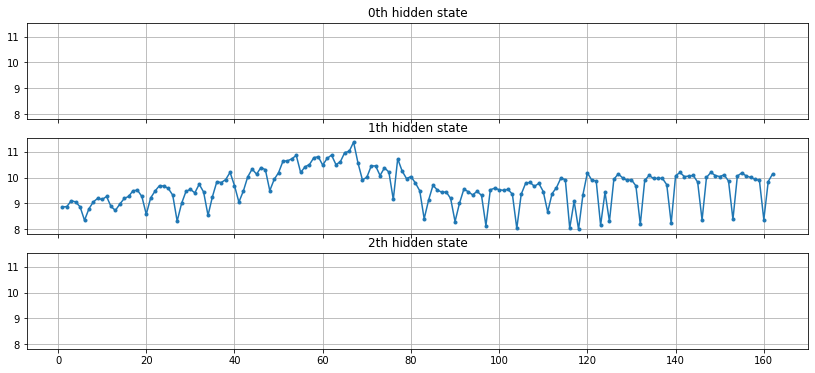
fig, axs = plt.subplots(model.n_components, sharex=True, sharey=True, figsize=(14, 6))
for i, ax in enumerate(axs):
mask = hidden_states == i
ax.plot(numpy.arange(0, ry.shape[0])[mask], ry[mask], ".-")
ax.set_title("{0}th hidden state".format(i))
ax.grid(True)
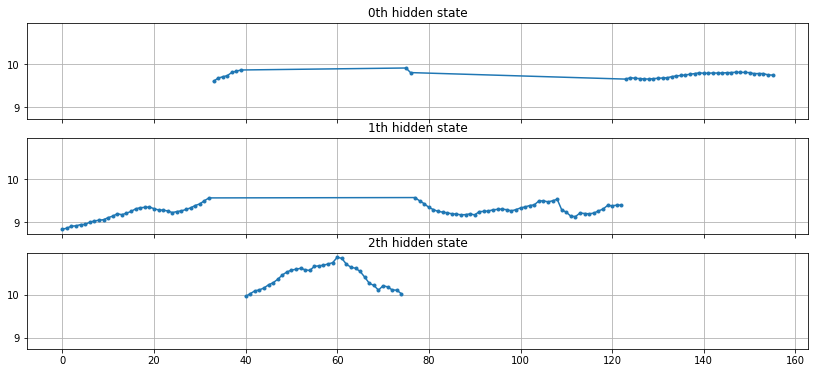
Si le modèle gaussien a l’air de fonctionner sur la série tendancielle (hors saisonnalité), il est plutôt utilisé pour segmenter selon la loi des résidus. Sur chaque segments, la série peut être approchée par une loi normale de moyenne et variance différentes.
model.means_, model.covars_
(array([[ 9.75077896],
[ 9.26327355],
[10.40300854]]),
array([[[0.00506213]],
[[0.02444731]],
[[0.07163662]]]))
ExponentialSmoothing#
Le lissage exponentiel consite à modéliser la série comme suit :
Ou :
Ou plus complexe avec Holt Winters:
from statsmodels.tsa.holtwinters import ExponentialSmoothing
fit1 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='add').fit(use_boxcox=True)
fit2 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='mul').fit(use_boxcox=True)
fit3 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='add', damped=True).fit(use_boxcox=True)
fit4 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='mul', damped=True).fit(use_boxcox=True)
c:python387_x64libsite-packagesstatsmodelstsaholtwintersmodel.py:427: FutureWarning: After 0.13 initialization must be handled at model creation warnings.warn( c:python387_x64libsite-packagesstatsmodelstsaholtwintersmodel.py:1112: FutureWarning: Setting use_boxcox during fit has been deprecated and will be removed after 0.13. It must be set during model initialization. warnings.warn( <ipython-input-26-0a978064fcad>:4: FutureWarning: the 'damped'' keyword is deprecated, use 'damped_trend' instead fit3 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='add', damped=True).fit(use_boxcox=True) <ipython-input-26-0a978064fcad>:5: FutureWarning: the 'damped'' keyword is deprecated, use 'damped_trend' instead fit4 = ExponentialSmoothing(y, seasonal_periods=7, trend='add', seasonal='mul', damped=True).fit(use_boxcox=True)
fit1.summary()
| Dep. Variable: | endog | No. Observations: | 162 |
|---|---|---|---|
| Model: | ExponentialSmoothing | SSE | 7985315446.470 |
| Optimized: | True | AIC | 2891.550 |
| Trend: | Additive | BIC | 2925.514 |
| Seasonal: | Additive | AICC | 2894.010 |
| Seasonal Periods: | 7 | Date: | Wed, 10 Feb 2021 |
| Box-Cox: | True | Time: | 23:00:39 |
| Box-Cox Coeff.: | 0.19415 |
| coeff | code | optimized | |
|---|---|---|---|
| smoothing_level | 0.2185487 | alpha | True |
| smoothing_trend | 0.1681336 | beta | True |
| smoothing_seasonal | 0.2405703 | gamma | True |
| initial_level | 29.375329 | l.0 | True |
| initial_trend | 0.1857434 | b.0 | True |
| initial_seasons.0 | -5.9555579 | s.0 | True |
| initial_seasons.1 | -5.4784169 | s.1 | True |
| initial_seasons.2 | -5.2732824 | s.2 | True |
| initial_seasons.3 | -5.0576235 | s.3 | True |
| initial_seasons.4 | -6.8353828 | s.4 | True |
| initial_seasons.5 | -10.316491 | s.5 | True |
| initial_seasons.6 | -7.6978206 | s.6 | True |
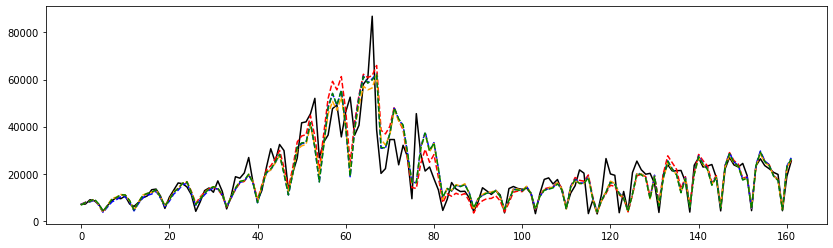
fig, ax = plt.subplots(1, 1, figsize=(14, 4))
ax.plot(y, 'b', color='black', label="Cas positifs")
ax.plot(fit1.fittedvalues, '--', color='red')
ax.plot(fit2.fittedvalues, '--', color='blue')
ax.plot(fit3.fittedvalues, '--', color='orange')
ax.plot(fit4.fittedvalues, '--', color='green');
Single Spectrum Analysis (SSA)#
Le module `pyts <>`__ ne fonctionnait pas pour moi. Je suis alors le notebook Single Spectrum Analysis (SSA).
def lagged_ts(serie, lag):
dim = serie.shape[0]
res = numpy.zeros((dim - lag + 1, lag))
for i in range(lag):
res[:, i] = serie[i:dim-lag+i+1]
return res
lagged_ts(y, 3)[:5]
array([[7017., 7157., 8975.],
[7157., 8975., 8550.],
[8975., 8550., 7071.],
[8550., 7071., 4203.],
[7071., 4203., 6544.]])
n_lag = 7
lag = lagged_ts(y, n_lag)
u, s, vh = svd(lag)
u.shape
(156, 156)
from numpy.linalg import svd
np = 6
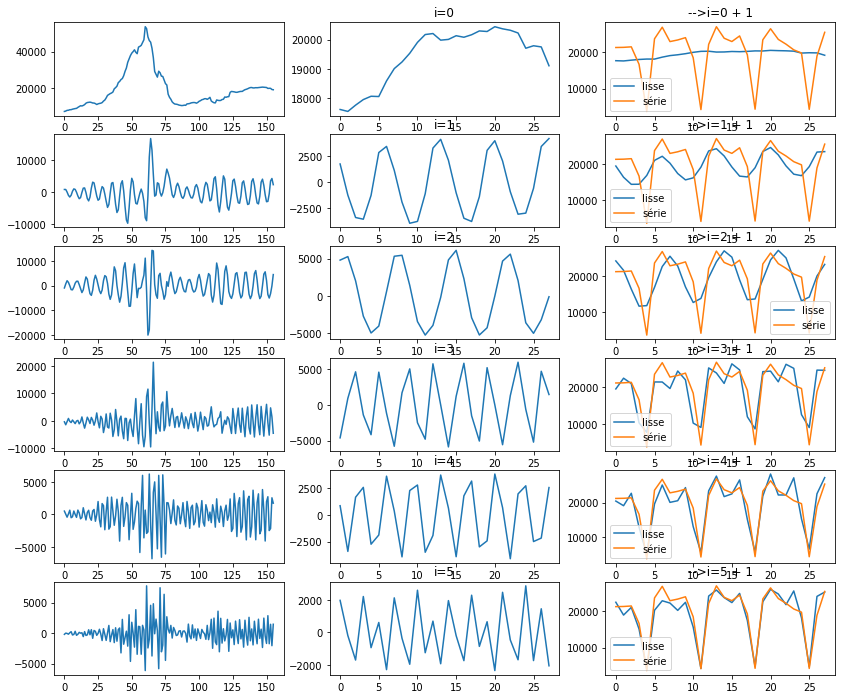
fig, ax = plt.subplots(np, 3, figsize=(14, np*2))
for n in range(np):
i = n
d = numpy.zeros((u.shape[0], n_lag))
d[i, i] = s[i]
X2 = u @ d @ vh
pos = 0 #X2.shape[1] - 1
# série reconstruites avec un axe
ax[n, 0].plot(X2[:,pos])
ax[n, 1].set_title("i=%d" % i)
ax[n, 1].plot(X2[-29:-1:,pos])
d = numpy.zeros((u.shape[0], n_lag))
d[:i+1, :i+1] = numpy.diag(s[:i+1])
X2 = u @ d @ vh
ax[n, 2].plot(X2[-29:-1,pos], label="lisse")
ax[n, 2].plot(y[-28:], label="série")
ax[n, 2].set_title("-->i=%d + 1" % i)
ax[n, 2].legend()
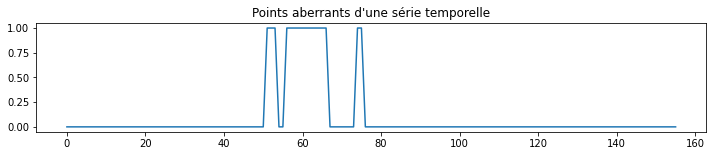
Points aberrants#
d = numpy.zeros((u.shape[0], n_lag))
for i in range(0, d.shape[1]):
d[i, i] = s[i]
X2 = u @ d @ vh
from sklearn.covariance import EllipticEnvelope
env = EllipticEnvelope(support_fraction=0.9)
env.fit(X2[:,:n_lag // 2])
out = env.predict(X2[:,:n_lag // 2])
fig, ax = plt.subplots(1, 1, figsize=(12,2))
ax.plot((1 - out)/2, "-")
ax.set_title("Points aberrants d'une série temporelle");
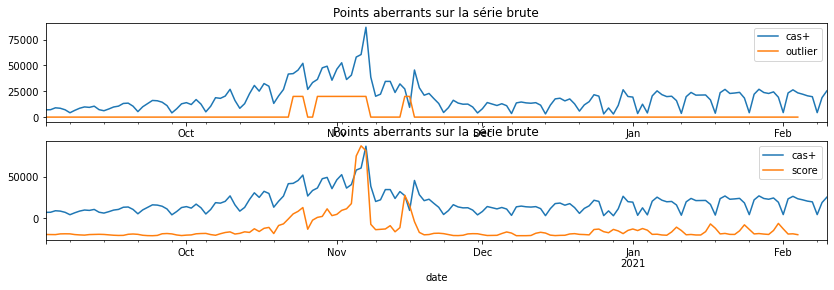
fig, ax = plt.subplots(2, 1, figsize=(14,4))
outp = env.decision_function(X2[:,:n_lag // 2])
dfy = DataFrame(data=y, index=dates, columns=["cas+"])
dfy['outlier'] = dfy['score'] = numpy.nan
dfy.iloc[:-n_lag+1, 1] = (1 - out)*1e4
dfy.iloc[:-n_lag+1, 2] = -outp*1e3
dfy[['cas+', 'outlier']].plot(ax=ax[0])
ax[0].set_title("Points aberrants sur la série brute");
dfy[['cas+', 'score']].plot(ax=ax[1])
ax[1].set_title("Points aberrants sur la série brute");